Em mais um tópico da coluna ‘Drops de Genômica, o oncologista André Murad (foto) explica como funciona a regulação da longevidade das proteínas pelo sistema ubiquitina-proteassoma. “As enzimas do metabolismo da ubiquitina foram identificadas como oncogenes ou supressores de tumor em vários tipos de câncer”, afirma. Confira.
Em mais um tópico da coluna ‘Drops de Genômica, o oncologista André Murad (foto) explica como funciona a regulação da longevidade das proteínas pelo sistema ubiquitina-proteassoma. “As enzimas do metabolismo da ubiquitina foram identificadas como oncogenes ou supressores de tumor em vários tipos de câncer”, afirma. Confira.
Por André Marcio Murad*
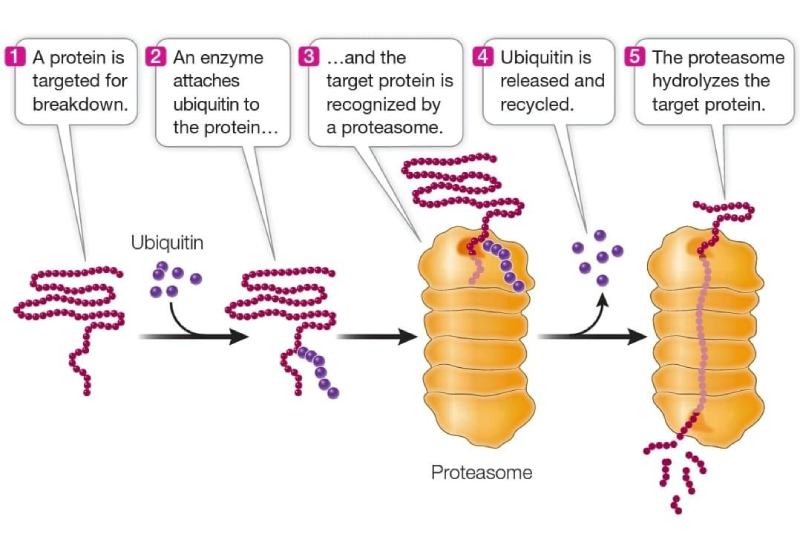
O conteúdo proteico de uma célula em um determinado momento é uma função tanto da síntese, quanto da degradação proteica. Certas proteínas podem ser alvo de destruição numa cadeia de eventos que começa quando uma enzima liga uma proteína de 76 aminoácidos chamada ubiquitina (assim chamada porque é onipresente e generalizada) a um resíduo de lisina da proteína a ser destruída.
Outras ubiquitinas se ligam à primária, formando uma cadeia de poliubiquitina. O complexo proteína-poliubiquitina liga-se então a um enorme complexo proteico denominado proteassoma. Ao entrar no proteassoma, a poliubiquitina é removida e a energia ATP é usada para desdobrar a proteína alvo. Três proteases diferentes digerem a proteína em pequenos peptídeos e aminoácidos.
Um proteassoma decompõe proteínas. As proteínas alvo de degradação estão ligadas à ubiquitina, que então liga a proteína alvo a um proteassoma, uma estrutura complexa onde as proteínas são digeridas por várias proteases poderosas.
Em virtude da sua capacidade de regular tanto a renovação proteica como as funções de sinalização não proteolítica, a conjugação da proteína ubiquitina tem sido implicada no controle de múltiplos processos celulares, incluindo localização de proteínas, controle do ciclo celular, regulação da transcrição, reparação de danos no DNA e endocitose. As enzimas do metabolismo da ubiquitina foram identificadas como oncogenes ou supressores de tumor em vários tipos de câncer.
Dado que o metabolismo da ubiquitina é governado por enzimas - E1, E2, E3, E4, deubiquitinases (DUBs) e o proteassoma - o sistema como um todo está maduro para a descoberta de alvos e medicamentos no câncer. Dos componentes do sistema ubiquitina/proteassoma, os E3s e DUBs podem reconhecer substratos com maior especificidade e são, portanto, de interesse fundamental como alvos potencialmente terapêuticos.