 Em mais um tópico da coluna Drops de Genômica, o oncologista André Murad (foto) aborda o passado, presente e futuro das tecnologias de sequenciamento genético do DNA. Confira.
Em mais um tópico da coluna Drops de Genômica, o oncologista André Murad (foto) aborda o passado, presente e futuro das tecnologias de sequenciamento genético do DNA. Confira.
O sequenciamento de DNA passou por avanços radicais desde que o primeiro método de sequenciamento Sanger foi introduzido em 1977. O sequenciamento Sanger faz uso de desoxinucleotídeos de terminação de cadeia adicionados aleatoriamente durante a síntese de DNA para gerar cadeias de DNA de comprimentos diferentes por um par de bases. São então separados em um gel de poliacrilamida e "lidos" da esquerda para a direita como um livro. Atualmente, existe uma grande variedade de métodos aprimorados com resultados rápidos e precisos.
Do método Sanger ao sequenciamento de molécula única
No final dos anos 1990 e 2000, o Projeto Genoma Humano promoveu melhorias no método Sanger mencionado anteriormente, que inclui o sequenciamento "shotgun" do genoma inteiro e métodos de sequenciamento de próxima geração (NGS). Ao quebrar grandes sequências de DNA e sequenciá-las simultaneamente de maneira massivamente paralela, os dados podem então ser gerados mais rapidamente, através de técnicas especiais de bioinformática. Com o aprimoramento desta tecnologia computacional, a sequência pode ser remontada e genomas inteiros passam a ser decodificados.
Empresas como Solexa e Life Technologies aprimoraram esses métodos mais uma vez, criando tecnologias de sequenciamento de última geração. No sequenciamento de próxima geração, os fragmentos de modelo de DNA são anexados a superfícies sólidas como lâminas de vidro ou microesferas para um manuseio mais automatizado e de alto rendimento. Algumas inovações químicas foram adicionadas no sequenciamento de segunda geração. Esses métodos foram desenvolvidos e ganharam popularidade na década de 2000.
Análise Genética Helicos
A Helicos Genetic Analysis Platform sequencia moléculas de DNA individuais ligadas a uma superfície plana à medida que são estendidas, análogos de nucleotídeos marcados com fluorescência, chamados de nucleotídeos terminadores de cadeia. Essa tecnologia ainda requer uma pausa após cada rodada de extensão para se ler a sequência, mas elimina a etapa de PCR (reação em cadeia da polimerase) necessária para tecnologias de próxima geração, como Illumina, Ion AmpliSeq da Thermo Fisher e Roche 454.
Sequenciamento em tempo real de molécula única
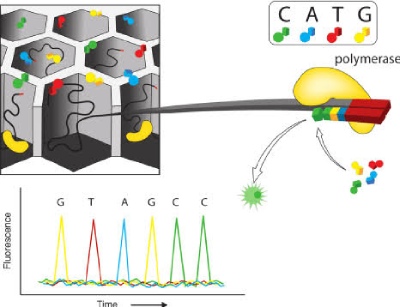
Mais recentemente, a Pacific Biosciences desenvolveu o método de sequenciamento em tempo real de molécula única (SMRT), que se baseia na observação direta da síntese de uma única fita de DNA. O sequenciamento SMRT utiliza uma câmara em escala nanométrica para isolar uma única fita de DNA e a DNA polimerase. A molécula de DNA polimerase é ancorada e então inundada com nucleotídeos marcados com fluorescência. Os nucleotídeos podem ser detectados à medida que se difundem para a fita de DNA ligada e são reconhecidos e incorporados pela DNA polimerase. O processo de difusão e extensão ocorre em milissegundos, o que significa que a DNA polimerase é adicionada várias bases por segundo. Como o corante fluorescente está ligado ao nucleotídeo por meio de uma cadeia de fosfato, ele se desconecta do nucleotídeo durante a reação da DNA polimerase, liberando-se da crescente cadeia de DNA.
*André Murad é diretor científico do Grupo Brasileiro de Oncologia de Precisão (GBOP), diretor clínico da Personal - Oncologia de Precisão e Personalizada, professor adjunto coordenador da Disciplina de Oncologia da Faculdade de Medicina da UFMG, e oncologista e oncogeneticista da CETTRO Oncologia (DF)