 O PCR (reação em cadeia da polimerase) digital é uma evolução das técnicas de PCR que permite uma quantificação absoluta, altamente sensível de ácidos nucleicos sem a necessidade de curva-padrão.
O PCR (reação em cadeia da polimerase) digital é uma evolução das técnicas de PCR que permite uma quantificação absoluta, altamente sensível de ácidos nucleicos sem a necessidade de curva-padrão.
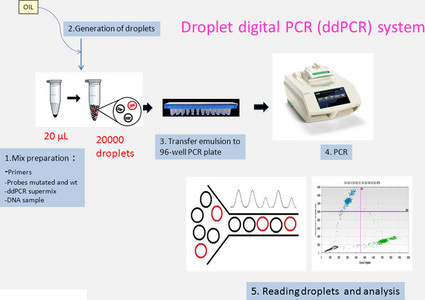
No sistema de PCR digital em gotas (dd-PCR), por exemplo, uma amostra é dividida em 20 mil gotas que, após a amplificação, aquelas contendo a sequência-alvo são detectadas por fluorescência e classificadas como positivas e gotas sem fluorescência são negativas.
A PCR digital melhora a sensibilidade da RT-PCR e permite a detecção de eventos raros, como mutações de um único nucleotídeo numa população de sequências de tipo selvagem. Na RT-PCR, o sinal de sequências de tipo selvagem pode dominar e obscurecer o sinal da sequência rara. Ao minimizar os efeitos da competição entre alvos, a PCR digital supera as dificuldades inerentes à amplificação de sequências raras, permitindo a quantificação absoluta sensível e precisa de ácidos nucleicos.
Um passo crítico na PCR digital é o particionamento da amostra. A amostra é preparada de uma forma semelhante à RT-PCR, mas é então separada em milhares de partições, cada uma contendo idealmente zero ou uma (ou, no máximo, algumas) molécula molde. Cada partição comporta-se como uma reação de PCR individual e, tal como a RT-PCR, são utilizadas sondas fluorescentes para identificar o DNA-alvo amplificado. Cada partição pode então ser prontamente analisada após amplificação para determinar se contém ou não a sequência-alvo.
As amostras contendo produto amplificado são consideradas positivas (1, fluorescentes) e as que não têm produto e, portanto, com pouca ou nenhuma fluorescência são negativas (0). A proporção de positivos para negativos em cada amostra é a base da quantificação.
Ao contrário da RT-PCR, a PCR digital não depende do número de ciclos de amplificação para determinar a quantidade inicial de ácido nucleico em cada amostra. Em vez disso, baseia-se no cálculo estatístico de Poisson para determinar a quantidade absoluta de DNA. O passo de particionamento da amostra de PCR digital, associado à análise de dados estatísticos de Poisson, permite mais precisão que os métodos tradicionais de PCR e RT-PCR.
A PCR digital é bem adequada para aplicações que requerem detecção de pequenas quantidades de ácido nucleico ou uma resolução precisa da quantidade de DNA-alvo entre amostras, por exemplo, detecção de sequências raras e análise de variação de número de cópias (CNV).
A PCR digital atinge maior alcance nas análises de ácidos nucleicos quando comparada a outros métodos, em uma série de aplicações:
– Biópsia líquida;
– variação do número de cópias (CNV);
– detecção de sequências raras;
– análise de expressão gênica e miRNA;
– análise de células isoladas single cell;
– detecção de patógenos;
– validação de biblioteca de NGS.